單細(xì)胞mRNA測序(scRNA-seq)允許分析異質(zhì)細(xì)胞群,提供解決各種生物和醫(yī)學(xué)問題的令人興奮的可能性��。最近開發(fā)了一系列方法��,因此有必要系統(tǒng)地比較它們的靈敏度���,準(zhǔn)確度,精度和成本效率��。
在這里����,來自Ludwig-Maximilians大學(xué)的研究人員已經(jīng)生成并分析了來自479只小鼠ES細(xì)胞和加標(biāo)對照的scRNA-seq數(shù)據(jù),這些對照用四種不同方法制備�����,每種方法分別進(jìn)行兩次重復(fù)。他們通過檢測基因的數(shù)量和它們捕獲加標(biāo)mRNA的效率��,通過將加標(biāo)mRNA濃度與估計的表達(dá)水平相關(guān)聯(lián)�����,通過功率模擬和方差分解的精確度以及它們的效率來比較它們的靈敏度���。達(dá)到一定數(shù)量電力的成本�。
實驗和計算管道的示意圖
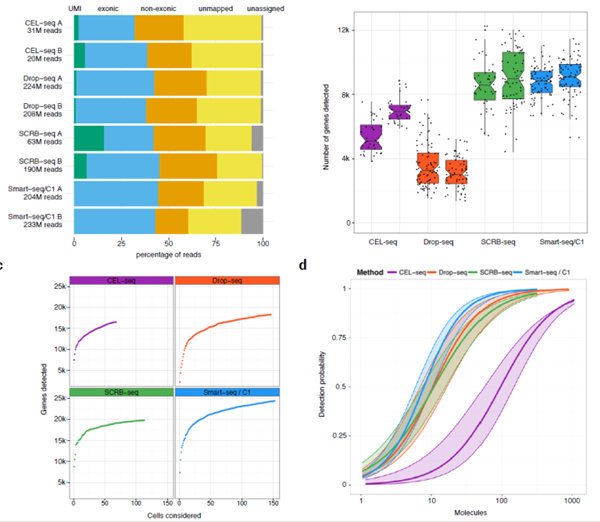
在2i / LIF和ERCC加標(biāo)RNA中培養(yǎng)的小鼠胚胎干細(xì)胞用于制備單細(xì)胞RNA-seq文庫�。這四種方法的不同之處在于獨特的分子標(biāo)識符序列(UMI)的存在和長度,允許鑒定cDNA擴(kuò)增過程中產(chǎn)生的讀數(shù)��。對于所有方法����,比較地進(jìn)行細(xì)胞的數(shù)據(jù)處理和子集化。每個方法和重復(fù)的最終細(xì)胞數(shù)用它們的平均測序深度顯示����。顏色代表比較的scRNA-seq方法:紫色 - CEL-seq,橙色 - Drop-seq��,綠色SCRB-seq,藍(lán)色 - Smart-seq并且在本研究中使用�����。
雖然所有方法的準(zhǔn)確度相似��,但研究人員發(fā)現(xiàn)微流體平臺上的Smart-seq是最敏感的方法���,CEL-seq是最精確的方法����,SCRB-seq和Drop-seq是最有效的方法�。該分析為四種可用的scRNA-seq方法的選擇提供了堅實的基礎(chǔ),并為未來的方法開發(fā)提供了基準(zhǔn)����。
scRNA-seq方法的靈敏度
(a)不能分配給細(xì)胞條形碼(灰色)的總讀數(shù)百分比,不能映射到小鼠基因組(黃色)����,
映射到外顯子(橙色)外部區(qū)域����,內(nèi)部外顯子(藍(lán)色)和攜帶獨特的UMI(綠色)�����。(b)總共讀數(shù)超過100,000的所有細(xì)胞中至少有一次讀數(shù)的基因數(shù)����。每個點代表一個細(xì)胞���,每個箱圖代表每個重復(fù)和方法的中位數(shù)���,第一和第三個四分位數(shù)。(c)添加更多細(xì)胞時檢測到的累積基因數(shù)����。(d)每種方法的靈敏度估計為檢測ERCC轉(zhuǎn)錄物的概率取決于其每個細(xì)胞的拷貝數(shù)。估計值的95%置信區(qū)間顯示為陰影區(qū)域�。
免責(zé)聲明:部分圖片來源于網(wǎng)絡(luò),如有侵權(quán)請聯(lián)系刪除�����!